Cuando entramos en LinkedIn, nos corresponde recomendar conectarse con personas que conocemos, fue porque estábamos en la misma universidad o coincidimos en la misma compañía. Sin embargo, siempre hay una sugerencia que nos sorprende. Por ejemplo, cuando el algoritmo recomienda nuestro cuñado, aunque trabajamos en campos completamente diferentes. Entonces, ¿cómo puede LinkedIn saber que "en la vida real" nos conocemos?
Los algoritmos de inteligencia artificial que promueven estas recomendaciones utilizan tecnología específica conocida como un gráfico de redes neuronales o redes neuronales gráficas, basadas en gráficos, estructuras formadas por nodos y bordes que las vinculan. En el contexto de las redes sociales como LinkedIn, puede generar un gráfico donde los nodos representan a cada usuario, mientras que los bordes corresponden a los enlaces entre ellos.
Estos algoritmos recopilan información de la vecindad inmediata de cada nodo, es decir, nuestros enlaces directos en LinkedIn. Luego agrupan esta información y la integran en el nodo original.
Por lo tanto, después de este proceso, cada perfil tiene una representación actualizada que refleja sus propios datos y los de sus redes más cercanas. Este proceso se puede realizar varias veces. Entonces, en otra iteración, cuando agregamos información de nuestros vecinos, a su vez agregará información de sus propios vecinos y, por lo tanto, tendremos información de otro entorno.
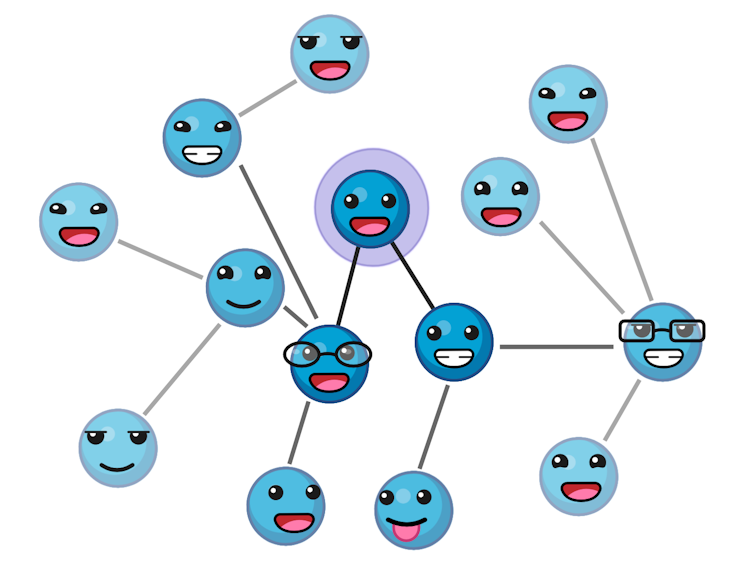
Un ejemplo de una red social. Lila Node representa nuestro perfil de LinkedIn. Desde su conexión directa (primeros vecindarios), así como los enlaces de estas personas, forman otro y tercer vecindario. М. Hernaez / Biorerer. Una red de enlaces
Por lo tanto, en estas redes, no solo las cosas no son solo nuestros datos personales, sino con quienes conectamos y conectamos nuestras conexiones. En la versión completa de los algoritmos de LinkedIn, como se usa en la práctica, no solo hay nodos que representan a los miembros de la red social, hay otros tipos de nodos, como empresas o publicaciones.
De esta manera, el algoritmo puede obtener información de nuestras conexiones personales y contenido que marcamos como favoritos o con el que nos comunicamos.
Por lo tanto, si alguien tiene a su hermana como relación y también dio las publicaciones que les gusta el hermano en lo que, AV, el algoritmo revela no solo para compartir intereses similares, sino que de alguna manera son de alguna manera de alguna manera.
¿Qué pasaría si pudiéramos usar este tipo de algoritmo de biomedicina?
Hoy, el desarrollo de un medicamento de cero es un procedimiento muy costoso, ambos en el tiempo también necesarios en las inversiones de capital. Muchas veces el proceso de descubrimiento se muestra como a la izquierda: al mismo tiempo, todos los candidatos posibles incluyen, después de diferentes fases de investigación, solo una para el examen clínico, que estará disponible y estará disponible para uso médico en la población.
Esto condujo al hecho de que en las últimas décadas, le preguntó a la configuración médica de Glory: no se les pide que diseñen un nuevo medicamento, sino que encuentren un nuevo uso para los medicamentos existentes.
Objetivos de proteína
En general, para el tratamiento de la enfermedad, nos centramos en el ataque a las proteínas responsables. Hay bases de datos públicas y muy distraíferas que contienen información sobre qué proteínas Ataque de drogas. En los últimos años, esta base de datos ha aumentado significativamente.
Como ejemplo, uno de los más utilizados, permaneció de 841 medicamentos aprobados en su primera versión (2006), en 2 751 en la última actualización (2024). Esta reciente disponibilidad de datos permite utilizar el uso de modelos complejos.
Entonces, como mencionamos anteriormente, podemos crear una red donde los medicamentos y las proteínas son nodos, y los bordes se interactúan entre ellos registrados en bases de datos. Una vez que tenemos una red, podemos aplicar un algoritmo similar: para cualquier proteína de información de drogas (bioquímica) que se agrega a través de enlaces conocidos.
Con esta información, el modelo puede decirnos las posibilidades de interacción en una proteína de drogas que a priori no está registrado en la base de datos. Los algoritmos pueden analizar efectivamente grandes cantidades de información. Después de eso, estas interacciones fueron confirmadas en el laboratorio, lo que permite ahorros y dinero.
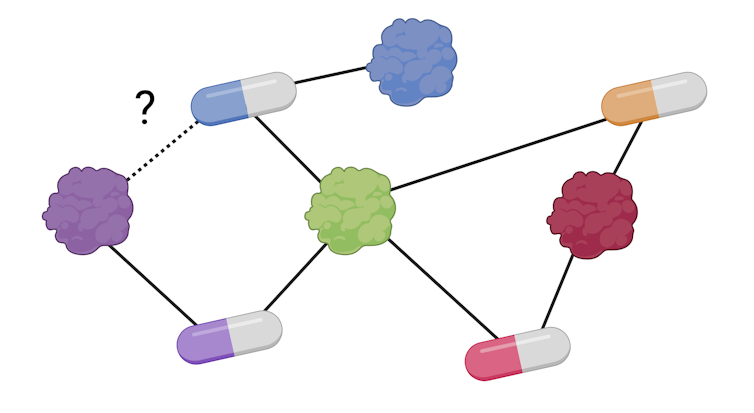
Una red de interacción de drogas de drogas. Las conexiones negras representan interacciones familiares. La firma de la pregunta en la línea discontinua indica la interacción de cuya existencia queremos confirmar. М. Hernaez / Biorrider nuestra contribución
En nuestro Laboratorio de Henea de Computer y Traduccional, Universidad de Navarra, seguimos esta idea para desarrollar Gennius, un modelo que considera la red entre medicina y proteínas. Con su aplicación, mejoramos los modelos existentes, especialmente en el momento de la ejecución: en solo una vez podemos evaluar unas 23,000 interacciones.
Sin embargo, aunque el modelo representa buenas oportunidades de pronóstico, todavía existe el borde de la mejora. Por ejemplo, uno de los desafíos ocurre durante la evaluación de posibles interacciones con moléculas que no son parte de la red o que es muy poco conocida. Aunque técnicamente es posible hacerlo, el modelo generalmente ofrece bajos resultados de confianza en estos casos.
Al guardar estos obstáculos y más investigaciones, estos modelos podrían desarrollarse en el futuro de acuerdo con los sistemas que ofrecen recomendaciones personalizadas para cada paciente.

0 Comentarios